精密健康在你的手掌

精密健康是健康的一种方法,考虑变化的基因,环境,和每个人的生活方式。由于医疗技术的进步,这是今天在这里。大量的数据被收集和分析管理我们的关怀,与数据源包括实验室测试,生物传感器,病人记录,医院的数据,等等。但结果可以缓慢的到来,等待测试和诊断之间可以几天或几周。
然而,最近突破实时基因组测序技术的发展,分析和诊断将提供个性化服务的新标准。
想象一个案件中,一个病人被承认一个诊所,一个简单的血液或者唾液测试管理。访问结束前,一个完整的诊断和个性化的治疗方案。在另一个场景中,一位外科医生正试图消除影响最小的肿瘤健康组织可以通过实时组织样本分析确认决定。最后,想象一个便携式病原体检测器可以提醒用户危险的大流行期间接触或疾病暴发。
这些和其他异象真正的关键将是一个手持设备,提供实时的基因组测序和分析病人的DNA或病原DNA或RNA。
基因测序技术的进步
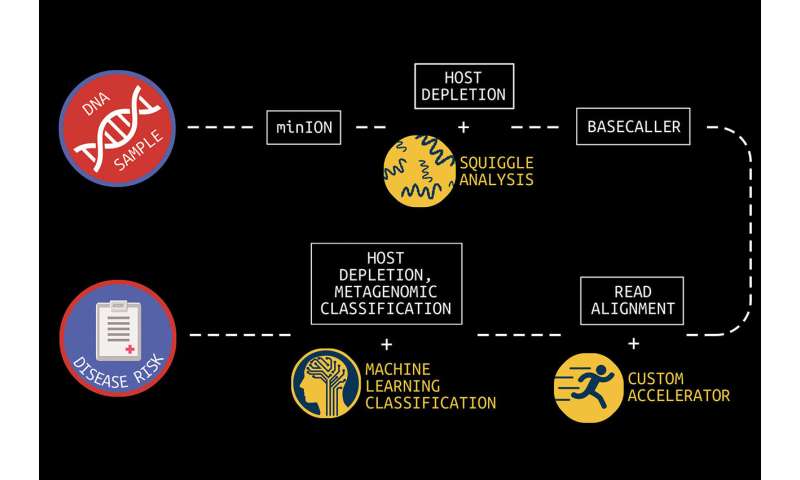
花费近30亿美元在2001年第一个人类基因组序列。今天,整个人类基因组测序成本低于1000美元,很快将达到约100美元。此外,第一代和第二代测序系统是大型,昂贵的,设计为批处理操作。结果采集标本后可用天或更多。但是新的、低成本的第三代测序系统现在存在,比如牛津纳米孔的奴才,可快速序列个别样品和适合你的手掌。
人类基因组是由超过三十亿个DNA碱基对。基因组序列,奴才雇佣小纳米孔收集样本划分为数十亿链,称为“读”。
“奴才手持测序是一个伟大的工具,能够快速测序生物数据,“说Reetuparna Das, CSE副教授。“化学样品,将DNA或RNA链,这些链成电子信号和序列,被称为“曲线”。However, it does not have the compute capability to analyze raw data in the field and quickly produce actionable results."
我们之间的所有,和实时诊断是一个计算系统,可以分析测序数据并提供治疗和治疗建议在病人离开办公室之前。
计算的挑战
在所谓的二次分析,计算系统的工作解释曲线作为DNA碱基对,这一过程被称为basecalling。一个碱基对本质上是一个DNA或RNA结构的地位。后,系统必须对齐参考基因组数据的读取数据,然后确定样本之间的变异和参考。人类基因组的变异数据是用来识别一种遗传性疾病标记。测序也用于识别病原体通过调整DNA或RNA链引用病原体数据库和使用宏基因组分类工具。
虽然这听起来简单,测序产生对GBs TBs的数据和精确的处理挑战是严峻的,因为,任务的复杂性和规模。密歇根大学的两个研究多学科小组正在研究方法来克服这个障碍。
教授和副教授Reetuparna Das Satish他,连同在电气和计算机工程教授大卫·布劳,是领导一个团队由美国国家科学基金会和卡恩基金会正在开发一个硬件/软件平台加速新一代基因组测序与关注病原体检测和早期癌症检测。在这方面,他们勾结内科医学、微生物学和免疫学副教授罗伯特·迪克森和卡尔Koschmann儿科助理教授,副教授以及詹娜Wiens,同时也是第二个研究团队的一部分。ob体育开户网址
第二小组,由卡恩基金会正在开发数据采集和机器学习技术来显著提高预测,在人口老龄化疾病的治疗和管理。这种努力的一个关键组件是使用机器学习速度宏基因组分析。
这个大规模的跨学科的努力是一个以色列技术研究所的研究人员之间的合作,魏兹曼科学研究所,密歇根大学。教授密歇根大学研究人员由贝琪·福克斯曼在公共卫生学院的流行病学。恩斯,他也是一个密歇根大学精密医疗主任,是一个Co-PI密歇根大学研究小组。
基因组测序的加速计算平台
布劳、Das和他的重点是大大加速和优化管道来处理数据的奴才。研究人员说,目标是减少所需的时间分析基因组测序的CPU小时几分钟内。
”意识到基因组测序的全部潜力,”达斯说,“计算能力需要增加数量级。”
问题是,那是不可能的在传统处理器路线图,额外的晶体管和核心在哪里更紧紧地挤在一个处理器的增量处理收益。添加额外的编程核心也不会解决这个问题。
“可持续增长的处理器性能只可能使用自定义层包括硬件,软件,和算法,”达斯说。
有很多领域的效率低下时产生的二次分析团队解决。
达斯说,第一是读一致性的过程,在此期间读取数据是基因组参考数据一致。读一致性由两个步骤组成:播种和种子扩展。

播种在参考基因组中发现一组候选位置,读可以对齐。可能的比赛被称为支安打的参考。在种子扩展阅读,参考字符串在热门职位匹配阅读。利用现有技术,这需要数以百计的CPU小时整个基因组。
播种,研究人员发现了一个巨大的内存带宽瓶颈。硬件/软件合作设计和开发了一种新的算法,数据结构和索引,交易掉内存容量内存带宽。然后建立一个定制的加速器,遍历新索引有效找到支安打和种子。播种算法已经发布开源软件和计划结合先进的校准软件Broad研究所和英特尔。
种子扩展,他们建立了一个心动阵列,将几百个周期使用近似串匹配匹配阅读和参考数据。
研究人员已经开发出一种定制ASIC来消除吞吐量瓶颈利用修剪算法优化DNA的对齐读取和候选基因突变,通过减少浮点计算43 x当真实的人体数据进行测试。
这些增强功能和其他人已经映射到自定义硬件。这包括种子扩展达到2.46米的加速器读取/秒,27 ~ 1800倍的性能改进和x小硅足迹而Xeon E5420处理器。
研究人员称,当高端56-core服务器上运行在亚马逊云,辅助分析工具需要大约6小时的全基因组测序。FPGA在亚马逊服务器,这降低了大约20分钟。当运行在研究者的定制硬件,处理时间是一分钟。
团队也开发了病原体检测技术来优化阅读过程。一是快速分析的开始阅读,以确定如果是主机或病原体的材料。如果是主机,其余的可以跳过阅读因为它只是感兴趣的病原体的材料。此外,研究人员通常能够完成这个宿主与病原体分化上使用机器学习花体数据,而不需要资源密集型basecalling。
微生物分析,提供更快的见解
当处理临床样本、快速数据处理管道输送的关键可行的见解。
“在临床样本,大部分的数据(有时多达90% -宿主DNA,而不是微生物DNA,”米拉Krishnamoorthy说,博士生和恩斯一起工作。“因此,现有的宏基因组分类工具商店很多有关该主机的信息,这可以计算效率低下。”
合作的一个研究小组在密西根州医学和公共卫生学院的恩斯和Krishnamoorthy正在in-silico机器学习方法主机损耗,或删除主机读取数据,这将成为一个Das的一部分,布劳,他是自定义硬件。他们的目标是消除所有的主机数据之前允许下游微生物分析只关注微生物分类数据。现有主机损耗方法是基于实验室和资源密集型的执行。
相比之下,Krishnamoorthy和恩斯的方法是计算,不依赖于大型参考数据库,而是基于卷积神经网络。需要输入读取输出的basecaller之后一系列的隆起和池步骤输出预测关于是否阅读属于主机。该方法提出了增加下游分析的效率,使微生物研究,有可能改变未来的医疗保健。
进一步探索