2014年7月22日特征
有前途的蛋白质:科学家使用光谱学和模拟开发新的药物发现工具
(欧宝娱乐地址Medical Xpress) - 在正在进行的追求设计新的有益分子或识别药学数据库中编目的潜在药物,批判性要求是确定一个配体(通常是一个调制器,或信号触发分子)与治疗蛋白结合。目前,大多数药物设计方案最有效的配体被选中 - 但以减少的靶蛋白质特异性成本。另一方面,这种小分子配体具有所谓的高Tc50.值,这意味着需要更大的药物来有效 - 因此可以被拒绝。然而,最近,Jawaharlal Nehru Nehru的科学家高级科学研究中心,印度组合了表面增强拉曼光谱(SERS)和分子动力学(MD)模拟,以识别调节剂结合的靶蛋白上的精确位置。(SERS是用于分析纳米级组合物的表面敏感技术,该技术可通过多达八个数量级增强正常拉曼信号; MD模拟原子和分子的物理运动,以细化蛋白质的三维结构和其他大分子。)
此外,研究人员通过测定抗高血压药物Felodipine与致癌基因的选择性结合(即导致肿瘤或肿瘤的发育)Aurora一种激酶人酶来证明了新的工具的疗效。此外,研究人员使用SERs选择性地抑制Aurora A的抗高血压性质,同时保留其抗癌性质 - 首次获取其知识 - 并使用分子对接研究和MD模拟证实这种选择性抑制。(分子对接是一种分子建模方法,其预测两种结合分子的优选取向,提供预测分子结合亲和力的能力。因此,科学家得出结论,他们的收购能力先天的的知识蛋白质结构表明,他们的新方案已经显示出非常强大的潜力,提供了在药物发现方面的进展,特别是新的高效力药物的开发。
Chandrabhas Narayana教授讨论了他和他的共同作者发表的论文国家科学院的诉讼程序用医疗Xp欧宝娱乐地址ress。关于使用SERs鉴定在治疗上重要的蛋白质上的小分子的结合位点,Narayana注意到SERS传统上用于检测具有高拉曼散射截面的小分子 - 即电场中分子的变形,这是它可以偏振的程度的函数。“然而,”Narayana告诉Medical Xpr欧宝娱乐地址ess“,使用SERS检测蛋白质等大分子,随后解释数据,是一般探索的具有挑战性的任务。因此,大多数治疗重要的蛋白质很少或没有结构信息。我们是第一个使用SERS调查这些蛋白质的小组。“
也就是说,Narayana指出,“最大的挑战是获得蛋白质的测量,其中表面电荷和表面疏水性决定纳米颗粒蛋白吸附 - 和标准拉曼光谱难以在这些治疗上的重要蛋白上进行,这主要是可能的影响蛋白质活动的纳米颗粒。另一个挑战,“他继续”是获取蛋白质结构信息,因为MD仿真和自动侦测很少预测这种系统的正确结构或小分子结合。“Autodock是一套自动分子对接工具,旨在预测小分子如何与已知的3D结构的受体结合。
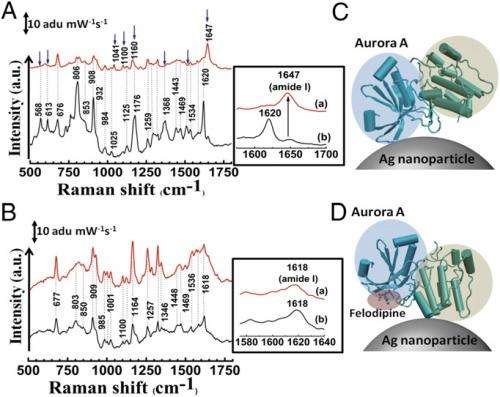
Narayana补充说,另一个挑战是理解蛋白质分子是如何附着在贵金属纳米颗粒表面的,后者是SERS现象的原因。(一种贵金属,例如金、银和铂,能够抵抗化学作用、腐蚀和酸。)“最重要的挑战是,蛋白质的表面增强拉曼光谱测量并不是一个完整的拉曼光谱,”他解释说,“但正如它的名字所表明的那样,焦点靠近纳米颗粒的表面。因此,解释不是很直接。然而,在进行SERS实验之前,对接研究产生了非常不同的位置——这是对接研究中常见的问题,因为有多种可能的基态。因此,对这些位点的突变研究从未给出期望的结果。”然而,通过使用SERS、分子对接和模拟,科学家们能够识别出小分子在蛋白质上的结合位点。
重要的是,科学家使用独特的表面结合模式实现了抑制。“表面结合模式是先前未开发的极光抑制模式,”Narayana告诉Medical Xpress。欧宝娱乐地址“当绑定发生时,抑制发生的事实离开从活性位点不仅有趣,而且还提供了另一种抑制模式。”研究人员从分子动力学模拟中了解了这种抑制的机制——表明极光A ATP结合口袋的变化。
Narayana注意到分子对接也在研究中发挥着重要作用,特别是在寻找重素分子可以结合的准确疏水袋中起作用。“这种技术 - 基于Lamarckian遗传算法 - 节省了大量的计算时间,并且已被广泛用于确定小分子与蛋白质的结合。(自动码克使用Lamarckian遗传算法作为几个搜索算法之一,以找到最佳构象的若干搜索算法之一具有最低的结合能量。)“停靠位置形成分子动力学模拟的初始点,其中考虑蛋白质柔韧性,并提供关于在与蛋白质结合的小分子中发生的结构变化的信息。”
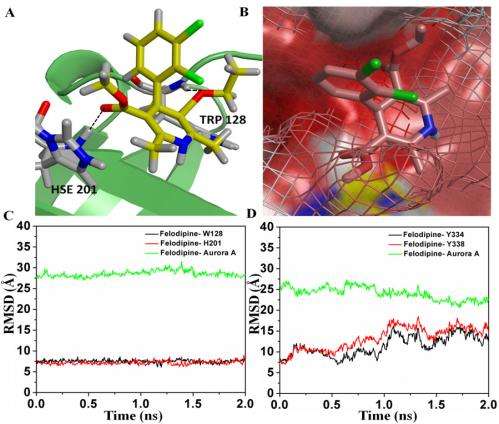
他补充说,由于有了快速高效的计算机,像蛋白质这样的大分子的分子动力学模拟成为可能。“我们与哈佛大学的汉斯·阿格伦教授的团队合作犹豫,瑞典,并且能够使用他们的计算设施。这分子动力学模拟这使得我们可以通过Aurora A结构的细微变化来了解其抑制非洛地平结合的机制。它也帮助我们验证非洛地平的对接位置。重要的是,我们已经使用了代码,我们可以在感兴趣的区域把蛋白质看作量子力学,在其他地方把粗颗粒看作粗颗粒——但再次强调,如果没有结构信息和使用SERS来验证所看到的效果,这是不可能的。”
在他们的论文中,科学家们表示,富县相关的化合物被视为抗肿瘤治疗剂。“一个人应该记住的是,作为一种抗高血压药物,我们必须首先衍生富沸性,使其失去其高血压控制,但保留了其抗癌性质,”Narayana指出。“此处的主要挑战是实现对利益目标的特殊性,以最少的偏离目标潜力。”(衍生化是在化学中使用的技术,其将化合物转化为称为衍生物的类似化学结构。)“使用这类化合物的主要障碍是实现最佳的治疗浓度,以治疗癌症而不阻挡维持至关重要的钙通道钙离子稳态。“换句话说,实现更高的治疗指数是在为特定病理情况设计小分子时需要考虑的重要因素之一。
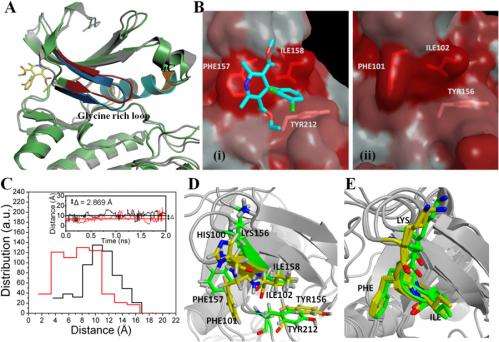
解决这些无数挑战的若干关键见解和创新是解决这些挑战的核心。“没有共同作者Tapas Kundu教授发现Felodipine是针对极光激酶选择性的,以及他的识别药物与极光A的互动的生化组成部分,我们的项目不会被取消,”Narayana告诉Medical Xpress。欧宝娱乐地址“此外,由于我们使用P300和CARM1等蛋白质的经验,我们意识到,虽然有可能对菌兵进行非常有趣的解释,但缺乏蛋白质的结构使得缺乏进一步的结构。因此,寻找蛋白质众所周知,谁的结构给了我们第一次见解,这对我们所取得的达到这一点来说,这将是一个好案例。蛋糕上的结冰是β薄片和α螺旋良好分开的事实。“(β-和α螺旋(或分别)是蛋白质二级结构-生物高聚物局部片段的三维形式,如蛋白质和核酸。)“多年来,我们对SERS技术进行了微调,以获得最佳信号,而无需纳米颗粒影响蛋白质活性或蛋白质在纳米颗粒表面的吸附。因此,我们已经开发出合适的纳米颗粒到蛋白质的浓度,压实纳米颗粒悬浊液以产生热点,并保持蛋白质周围的pH值,使其不会变性。”
Narayana注意到各种蛋白质的适当发达的SERS方法可用于快速筛选可能的配体,从而导致一系列治疗应用。“在Felodipine,其衍生物和仿制药物上进行的Sers表明,没有衍生物和通用药物扰乱在Aurora激酶上工作中的分子疏水 - 亲水性距离,尺寸或空间障碍物”Narayana说明“,”事实并不显示与Felodipine类似的任何变化。“(当分子内的大尺寸基团中,发生静透障碍阻止在具有较小群体的相关分子中观察到的化学反应。)“这告诉我们,如果我们在引入药物后看蛋白质的具体变化,第一次测试是研究其抑制作用或激活特性。为了成功,我们需要完全理解Sers Tending。“
接下来,科学家们将重点放在两个主要步骤上:利用这种技术来发现药物,以及研究蛋白质生物学中其他具有挑战性的问题,这些问题的结构变化将有助于了解蛋白质活性。
•药物设计/发现:
- 快速筛选的高吞吐量过程
-优化蛋白质目标阵列研究平台;在SERS活性纳米颗粒上使用合适的封盖剂;而纳米粒子功能化(即使用表面涂层来确定纳米粒子的性质)可以控制与特定蛋白区域的结合
•蛋白质结构研究:
- 促进人类蛋白质组研究
- 调查由带正电荷的离子驱动的蛋白质结构变化,或阳离子例如,遗传转录共活化或限制性内切酶(通过与激活物或转录因子结合而分别增加或减少基因表达的蛋白质)
Narayana还确定了研究人员可能考虑发展的其他一些创新 - 具体而言,更深入地了解尖端增强拉曼光谱(TERS) - 一种扫描探针显微镜技术,将拉曼光谱能力与原子力显微镜的空间分辨率相结合(<30 nm);体内Sers活性纳米颗粒的应用;为纳米颗粒吸附的蛋白质剪裁,靠近感兴趣的蛋白质遗址而不损失蛋白质活性。
总之,Narayana指出,还有其他研究领域可能会受益于他们的研究。这些包括筛选潜力小分子来自各种疾病的药物文库;合理的药物设计与有关现场有关的密切相关目标;设计正交成像的标签,而不会通过与独特的表面袋结合而不影响催化活性;了解基于C-肽的药物的特定相互作用;和体内蛋白质成像。
进一步探索
©2014医疗欧宝娱乐地址Xpress




.jpg)





用户评论